 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2012 年, 第 10 卷, 第 12 篇 doi: 10.5376/mpb.cn.2012.10.0012
收稿日期: 2011年12月16日 接受日期: 2012年02月24日 发表日期: 2012年04月26日
引用格式(中文):
任民等, 2012, dbMarker:基于云计算的分子标记管理系统, 分子植物育种(online) Vol.10 No.12 pp.1092-1096 (doi: 10.5376/mpb.cn.2012.10.0012)
引用格式(英文):
Ren et al., 2012, dbMarker: Molecular Marker Management System Based on Cloud Computing, Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding) Vol.10 No.12 pp.1092-1096 (doi: 10.5376/mpb.cn.2012.10.0012)
为了方便研究人员管理海量增长的分子标记信息数据,本研究采用Google App Engine云计算平台开发了dbMarker分子标记管理系统。利用该系统可以通过引物名称、前后引物序列等基本信息查询相关引物,同时还可对退火温度、扩增长度、引物所在染色体等设置查询条件检索符合用户要求的引物。此外,该系统还具备完善的数据上传更新功能,能够实现标记信息的安全稳定存储。dbMarker分子标记管理系统功能完备、界面友好、操作便捷,能够满足当前高通量研究手段对信息管理的要求。
自PCR技术发明以来,基于PCR的分子标记技术相继出现(Williams et al., 1990; Tautz, 1989; Zietkiewicz et al, 1994),随着分子标记的广泛应用和研究深度的不断增加,现已形成了以酶切、PCR和测序为基础的3大类几十种分子标记,一些常用分子标记的位点少则几千,多则如SNP在百万以上(Rodríguez-Suárez et al., 2011; Yu et al., 2011)。因此,单纯依靠人工已无法有效地进行各类标记信息的存储和管理。当前众多生物信息学网站均开发了标记信息管理系统(Sherry et al., 2001; Huala et al, 2001; Kim et al., 2008; Kim et al., 2009; Galperin and Fernández-Suárez, 2011; Sayers et al., 2011),在方便用户查询的同时也便于数据的收集和管理(表1)。然而面向实验室和个人的分子标记管理系统目前未见报道,随着研究的深入和高通量分析手段的普及,在研究分析过程中用到的标记的数量亦在迅速增加,相关数据分析、保存和整理急需由人工向信息化转变,故开发一个面向实验室和个人的分子标记管理系统就显得十分必要。本软件的研发正是在此背景下,在充分满足数据存储的条件下,还从实际应用和专业的角度出发有针对性的设计了用户查询功能。
|
表1文中所引数据库的分子标记查询网址 Table 1 The websites of molecular marker querying for the databases cited in this study |
1系统架构
dbMarker是基于云计算的信息管理系统,程序架构设计遵照了MVC模式,由模型、视图和控制器3组核心部件组成。模型负责执行分子标记数据的存储、更新和查询,视图负责数据展示和定义操作方式,是一组HTML格式网页,包括了系统首页面、数据查询页面、查询结果页面、数据上传和更新页面以及系统后台控制页面等;控制器负责响应用户发送的请求和选择业务处理逻辑,并更新视图以将查询结果反馈给用户(图1)。
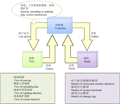 图1 dbMarker软件的MVC系统架构 Figure 1 System architecture and MVC design model of dbMarker software |
2界面设计
该软件的用户界面由一系列HTML页面组成,包括系统主页、查询、数据上传和更新以及系统维护等几组页面。
2.1系统主页
主页面如图2所示,最上方是系统功能导航栏,提供了数据查询、数据上传、数据更新和系统维护等软件主要功能的访问链接。其下是快速查询通道,以方便用户通过标记的名称或序列进行快速查询。再下方展示了已存储标记的一些统计信息,左侧的表格列出了数据库中字段的类型和含义,右侧实时生成的饼图直观的显示了数据库中的引物类型和数量。系统页面最下方为更新日志,当用户上传或更新了标记数据之后,可以撰写日志,以方便日后的查阅和使用。
 图2 dbMarker系统首页 Figure 2 The home page of dbMarker |
2.2查询页面
查询页面包括了提交检索信息的用户查询界面和返回查询结果的系统返回页面。其用户查询界面是一个由单选按钮、输入框、标签和提交按钮等元素组成的表单。用户提交查询信息后,系统以表格的形式返回查询结果,表格上方列有该次查询的条件。查询结果表格(图3)中英文单词或缩写的含义依次为:Name代表引物名称;Fwd和Res分别代表前后引物序列;Ftm、Rtm、Mtm和Dtm依次表示前引物TM值、后引物TM值、前后引物TM值的平均值和前后引物TM相差的绝对值;Fgc和Rgc分别表示前后引物的GC含量;Size代表扩增产物的长度,未知用“0”表示;Chro代表引物所在染色体的编号,未知用“0”表示;Type代表引物类型;Note为备注。
|
图3 返回用户查询结果的表格 Figure 3 The form showing query result to users |
2.3数据上传与更新页面
数据上传页面分为批量上传页面和逐条上传页面,数据更新页面也分为批量更新页面和逐项更新页面。批量上传页面和批量更新页面采用了相似的表单布局,均由密码框和输入标记数据的多行文本框组成,下方为提交按钮。逐条上传页面中每个信息字段对应一个文本框,标记类型字段为一个列表框,输入的内容仅能从列表框中选择。逐项更新页面由一个文本框输入待修改的引物名称,其下由一个列表框和文本框组合在一起供用户选择修改字段和输入更新信息。
3数据查询
dbMarker提供了两种查询方式:基本方式和快速方式。
基本方式:即在查询页面内执行查询操作,该方式提供了全面的查询方法。可在引物类型、引物名称、前后引物序列、染色体、扩增产物长度、平均退火温度、退火温度相差等字段设置查询条件,可几个字段联合组成查询条件,各个查询条件之间的逻辑关系为“与(AND)”。在需要设置条件的字段内输入查询条件,留空即为不限制该字段,但查询时必须选择一种引物类型。引物名称、前后引序列的输入内容为文本信息,染色体和扩增产物长度只接受正整数,平均退火温度和退火温度相差可接受小数(大于0)。在设置扩增产物长度、平均退火温度和退火温度相差字段时必须同时输入上限和下限。
快速方式:即利用主页的快速查询通道,直接输入引物的名称或序列,然后按回车键开始查询。针对用户已经知道引物的名称或者前后引物序列,欲查看该引物其他信息的情况。查询时dbMarker自动在引物名称和前后引物序列字段内检索用户提交的信息,其逻辑关系为“或(OR)”。
4数据上传或更新
批量上传数据或更新数据时,需按如下格式整理待上传或更新的数据信息:“Name, Fwd, Res, Ftm, Rtm, Fgc, Rgc, len, Chro, Type, Note”(格式不包含引号),字段之间用半角状态的逗号分隔,输入完一条记录后打回车键输入下一条记录。例如某标记的名称为mk1,前引物序列(5'-3')为AGACCTCATCAACAACCATCCA,后引物序列(5'-3')为GCTCCAGGGCACGCTCTTCT,前引物的退火温度为52℃,后引物的退火温度为57℃,前引物GC含量48%,后引物GC含量65%,扩增产物长度418 bp,位于1号染色体,标记的类型为ILP,备注无(NA)。批量上传或更新时应将该引物的数据整理为“mk1, AGACCTCATCAACAACCATCCA, GCTCCAGGGCACGCTCTTCT, 52, 57, 45, 65, 418, 1, ILP, NA”。然后输入到批量上传或更新页面相应文本框中,单击提交按钮后开始上传或更新;逐条上传数据时,需在逐条上传引物页面内按照提示信息,逐项输入相应的内容,其中引物类型从下拉列表中选择,完整填写表单后点击提交数据按钮完成该引物的数据上传;仅需要修改某个引物的个别记录时,可以采用逐项更新的方式。首先在逐项更新页面输入待更新引物的名称,然后从下拉列表中选择要更新的字段,并输入新的字段信息,最后点击提交按钮完成数据更新。
5讨论
云计算作为一种新兴的基于互联网的计算模式,实现了本地计算机或远程计算机的网络化协同工作,并且在生物信息学研究中得到了越来越广泛的应用(Dudley and Butte, 2010; Langmead et al., 2009)。2008年4月Google公司推出了一项名为Google App Engine的云计算服务,该云计算服务可让开发者更加专注于产品的研发,提高开发效率,降低技术风险。dbMarke正是基于该平台的云计算应用,实现了大量分子标记信息的便捷存储和查询。相比传统的保存方式(如利用EXCEL表格),效率更高,而且查询更加方便,能够满足当前高通量研究手段对海量信息处理的要求。与当前大型生物信息学网站的后台数据管理系统相比,dbMarker更加专注实验室和个人级别的分子标记信息存储和管理。首先,dbMarker软件起源于本研究实验室日常分子标记的管理和应用,在人工管理已经无法适应,而大型公共生物信息数据库又无法与日常研究工作紧系衔接的情况下,dbMarker的设计目标就定位于弥补上述缺口;其次,该软件的体积非常小巧,源文件只有30多K,而且Google App Engine平台为软件的发布和使用提供了极大的便利性,更不需要用户进行硬件方面的投入,大大降低了应用难度和技术风险。目前,该软件已经获得国家版权局授予的软件著作权保护登记(2010SR059772),并在Google代码网站上设立了项目托管,访问地址为:http://code.google.com/p/dbmarker/。用户可以通过以上地址了解技术信息,下载软件包以及源代码。
作者贡献
任民、盖红梅及蒋彩虹是本研究的实验设计和实验研究的执行人;任民负责系统架构和软件开发;盖红梅和蒋彩虹负责系统测试,任民、盖红梅完成了论文初稿的写作;王志德是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究由中国农业科学院烟草研究所所长基金“应用选择牵连效应发掘烟草基因组保守位点”、中央级公益性科研院所基本科研业务费专项(0032010038)和青岛市科技计划基础研究项目(10-3-4-14-1-jch)共同资助。
参考文献
Bombarely A., Menda N., Tecle I.Y., Buels R.M., Strickler S., Fischer-York T., Pujar A., Leto J., Gosselin J., and Mueller L.A., 2011, The sol genomics network (solgenomics.net): growing tomatoes using perl, Nucleic Acids Res., 39(Database issue): D1149-D1155
Dudley J.T. and Butte A.J., 2010, In silico research in the era of cloud computing, Nature biotechnology, 28(11): 1181-1185
http://dx.doi.org/10.1038/nbt1110-1181 PMid:21057489
Galperin M.Y., and Fernández-Suárez X.M., 2011, The 2012 nucleic acids research database issue and the online molecular biology database collection, Nucleic Acids Res., 40(D1): D1-D8
Huala E., Dickerman A.W., Garcia-Hernandez M., Weems D., Reiser L., LaFond F., Hanley D., Kiphart D., Zhuang JM., Huang W., Mueller L.A., Bhattacharyya D., Bhaya D., Sobral B.W., Beavis W., Meinke D.W., Town C.D., Somerville C., and Rhee S.Y., 2001, The arabidopsis information resource (TAIR): a comprehensive database and web-based information retrieval, analysis, and visualization system for a model plant, Nucleic Acids Res., 29(1): 102-105
http://dx.doi.org/10.1093/nar/29.1.102 PMid:11125061 PMCid:29827
Kim C.K., Kim J.S., Lee G.S., Park B.S., and Hahn J.H., 2008, PlantGM: a database for genetic markers in rice (Oryza sativa) and Chinese cabbage (Brassica rapa), Bioinformation, 3(2): 61-62
http://dx.doi.org/10.6026/97320630003061 PMid:19238232 PMCid:2637951
Kim C.K., Yoon U.H., Lee G.S., Lee H.K., Kim Y.H., and Hahn J.H., 2009, Rice genetic marker database: An identification of single nucleotide polymorphism (SNP) and quantitative trait loci (QTL) markers, African Journal of Biotechnology, 8(13): 2963-2967
Langmead B., Schatz M.C., Lin J., Pop M., and Salzberg S.L., 2009, Searching for SNPs with cloud computing, Genome Biology, 10(11): R134
http://dx.doi.org/10.1186/gb-2009-10-11-r134 PMid:19930550 PMCid:3091327
Rodríguez-Suárez C., Giménez M.J., Gutiérrez N., Avila C.M., Machado A., Huttner E., Ramírez M.C., Martín A.C., Castillo A., Kilian A., Martín A., and Atienza S.G., 2011, Development of wild barley (Hordeum chilense)-derived DArT markers and their use into genetic and physical mapping, Theor. Appl. Genet., 124: 713-722
http://dx.doi.org/10.1007/s00122-011-1741-2 PMid:22048641
Sayers E.W., Barrett T., Benson D.A., Bolton E., Bryant S.H., Canese K., Chetvernin V., Church D.M., Dicuccio M., Federhen S., Feolo M., Fingerman I.M., Geer L.Y., Helmberg W., Kapustin Y., Krasnov S., Landsman D., Lipman D.J., Lu Z., Madden T.L., Madej T., Maglott D.R., Marchler-Bauer A., Miller V., Karsch-Mizrachi I., Ostell J., Panchenko A., Phan L., Pruitt K.D., Schuler G.D., Sequeira E., Sherry S.T., Shumway M., Sirotkin K., Slotta D., Souvorov A., Starchenko G., Tatusova T.A., Wagner L., Wang Y., Wilbur W.J., Yaschenko E., and Ye J., 2011, Database resources of the National Center for Biotechnology Information, Nucleic Acids Res., 40(Database issue): D13-D25
Sherry S.T., Ward M.H., Kholodov M., Baker J., Phan L., Smigielski E.M., and Sirotkin K., 2001, dbSNP: the NCBI database of genetic variation, Nucleic Acids Res., 29(1): 308-311
http://dx.doi.org/10.1093/nar/29.1.308 PMid:11125122 PMCid:29783
Tautz D, 1989, Hypervariability of simple sequences as a general source for polymorphic DNA markers, Nucleic Acids Res., 17(16): 6463-6471
http://dx.doi.org/10.1093/nar/17.16.6463 PMid:2780284 PMCid:318341
Williams J.G.K., Kubelik A.R., Livak K.J., Rafalskil J.A., and Tingeyl S.V., 1990, DNA polymorphisms amplified by arbitrary primers are useful as genetic markers, Nucleic Acids Res., 18(22): 6531-6535 http://dx.doi.org/10.1093/nar/18.22.6531 PMid:1979162 PMCid:332606
Yu H.H., Xie W.B., Wang J., Xing Y.Z., Xu C.G., Li X.H., Xiao J.H., and Zhang Q.F., 2011, Gains in QTL Detection Using an Ultra-High Density SNP Map Based on Population Sequencing Relative to Traditional RFLP/SSR Markers, PLoS One, 6(3): e17595
http://dx.doi.org/10.1371/annotation/f2eb75fb-ae22-4a57-b828-1506aa506c6d
http://dx.doi.org/10.1371/journal.pone.0017595 PMid:21390234 PMCid:3048400
Zietkiewicz E., Rafalski A., and Labuda D., 1994, Genome Fingerprinting by Simple Sequence Repeat (SSR)-Anchored Polymerase Chain Reaction Amplification, Genomics, 20(2): 176-183 http://dx.doi.org/10.1006/geno.1994.1151 PMid:8020964